Oppgave 3
Bioinformatikk TES 8047
Melinda K. Christensen
Polymerase Kjede Reaksjon
Primer Konstruksjon
Introduksjon
Øvelsen gikk ut på å danne seg et oversikt på PCR (Polymerase Chain Reaction) og spesielt primer konstruksjon ved hjelp av aktuell software tilgjengelig på internett.
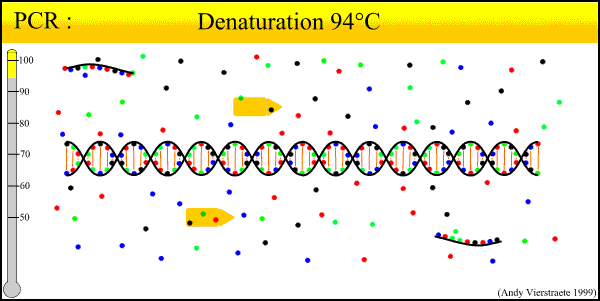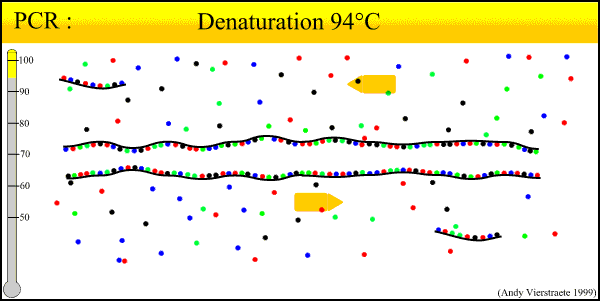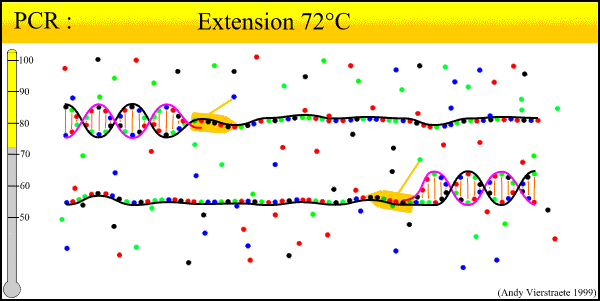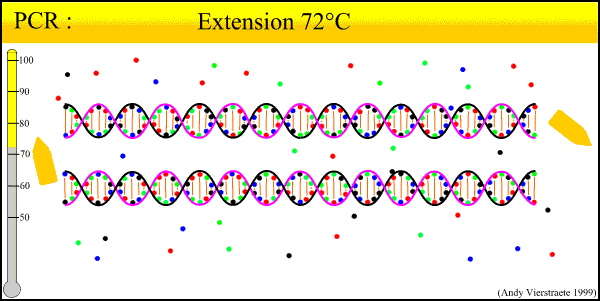
PCR
Polymerase Chain Reaction er en metode for amplifisering av et spesifikt område av DNA, slik at man kan produsere nok DNA til å teste. Denne teknikken kan benyttes for å identifisere bl.a. sykdomsfremkallende virus/bakterie, døde personer, eller personer mistenkt av en forbrytelse. Polymerase kjedereaksjon ble utviklet av Kary Mullis i 1985. Det er en syklisk prosess der en DNA sekvens fordobles i hver syklus. For å kunne benytte PCR teknikken må man kjenne den nøyaktige sekvensen som ligger til venstre og høyre for den biten man ønsker å amplifisere. Man utnytter det faktum at alle organismer har DNA sekvenser (gener) som er unike til den organismen.
Amplifiseringen skjer ved simultan primer forlengelse av komplementære DNA strenger.
DNA ( deoksyribosenukleinsyre) er arvematerialet i de fleste levende organismer. DNA’s stabile tilstand er en dobbelheliks som består av to antiparallel løpende DNA-enkeltstrenger. Enkeltstrengene er sukkermolekyler (deoksyribose) som er forbundet gjennom fosfatgrupper. En base sitter på hver deoksyribose molekyl og det er hydrogenbindinger mellom basene som binder to komplementære DNA-strenger sammen. Basene er cytosin (C) og guanin (G), som holdes sammen av 3 hydrogenbindinger og adenin (A) og tymin (T), som holdes sammen av 2 hydrogenbindinger. Det er denne forskjellen i bindingsenergi som må tas hensyn til i konstruksjon av primerpar for utførselen av PCR metoden.
Polymerase er enzymet som katalyserer dannelse av en DNA dobbelstreng. Enzymet benytter seg av en allerede eksisterende DNA enkelstreng som templat og setter inn komplementære nukleotider. dATP, dGTP, dCTP og dTTP (kalles dNTP’er) benyttes som substrat . Den komplementære DNA-strengen vokser i 5´® 3´ retning i DNA-syntese. For å kunne starte prosessen trengs en primer. Primeren er et lite DNA-stykke som er bundet til DNA-templatet. Primeren er altså start-punktet for replikasjon. PCR metoden hadde begrenset bruksområde inntil man fant at DNA polymeraser fra varmestabile organismer kunne benyttes.

Bildet er lånt fra Http://allserv.rug.ac.be/~avierstr/principles/pcr.html
I PCR metoden amplifiserer man en del av genet som man ønsker å påvise. Målet med amplifiseringen kan dekke et bredt spekter, bl.a.
Startmaterialet for PCR er en DNA matrise, varmeresistent DNA polymerase, dNTP’er, et passende primerpar samt buffer og en MgCl2 løsning. Mg2+ er nødvendig som koenzym.
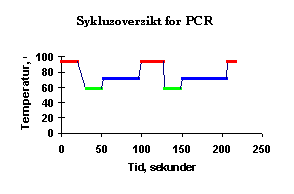
En PCR syklus består som regel av følgende tre trinn:
------
Representerer 1. Trinn------
Representerer 2. Trinn------
Representerer 3. Trinn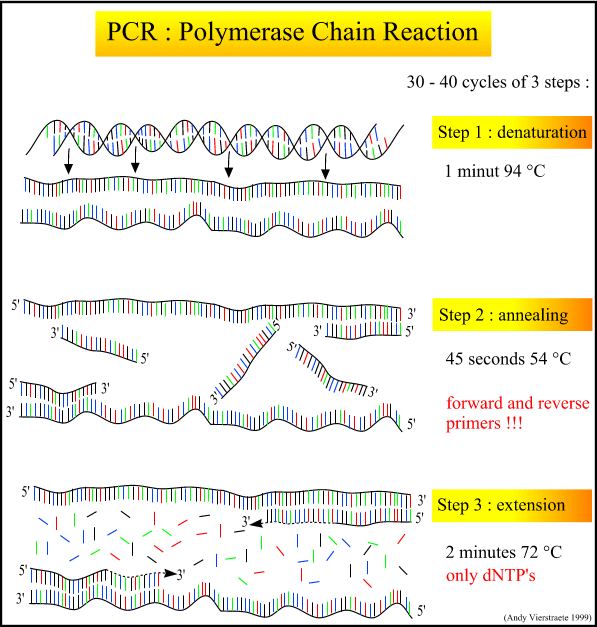
Bildet er lånt fra Http://allserv.rug.ac.be/~avierstr/principles/pcr.html
Som sagt tidligere fordobles DNA sekvensen i hver syklus. Etter 4. runde (syklus) formeres de to komplementære genstykkene,
eksponensielt. Det er denne sekvens man kaller målsekvensen eller amplikonet. Man har følgende algoritme for antall av enkel-streng amplikoner tilstede etter n-sykluser.
# amplikoner = 2 * (2n-n-1)
![]()
![]()
I produktblandingen har man også amplikoner som ikke er avgrenset av primere:
![]()
![]()
Disse vokser lineært med antall sykluser.
Total # ikke-avgrenset amplikoner = 2n
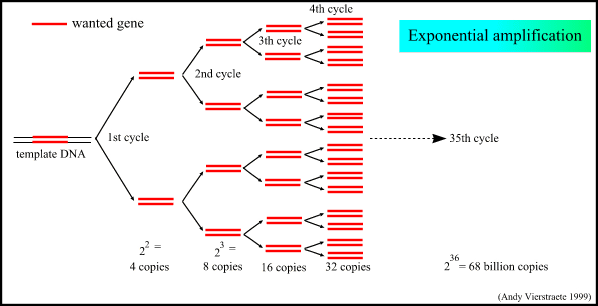
Bildet er lånt fra Http://allserv.rug.ac.be/¨avierstr/principles/pcr.html
Komponentene til PCR
Polymeraser
Polymeraser har, i vanlige celler en optimal arbeidstemperatur på ca. 37˚C og brytes ned ved ca. .Funn av varmestabile polymeraser har revolusjonert arbeid i amplifikasjon av DNA. Varmestabil polymerase kan motstå de temperaturer som er nødvendig for å lime på primeren slik at replikasjonen kan skje. Disse varmestabile enzymer ekstraheres enten direkte fra organismer som holder til i varmekilder eller fremstilles genteknologisk. Thermus aquatic (Taq) og Thermus thermophilus (Tth) er to av de forskjellige organismer som benyttes. De forskjellige polymeraser har ulik halveringstid, prosessivitet, arbeider med ulik hastighet og kan også ha en reverstranskriptase aktivitet. Det siste gjør at man er i stand til å lage cDNA fra RNA/mRNA. At polymerasene varierer så mye med hensyn til disse egenskapene gjør at man må være forsiktig i valg av polymerase type avhengig av bl.a. egenskapene til DNA matrisen man jobber med og lengden av amplikonet.
Substrat
Deoksynukleosidtrifosfater (dNTP’er) er nødvendig for energi og syntese av DNA. dNTP’er fås i handelen enten som fire separate løsninger (10mM) eller allerede blandet. Både dNTP’er og primer bør være med i stort overskudd slik at syntesetrinnet kan kjøres i syklus ved at det nylaget DNA denatureres ved oppvarming.
Buffer og MgCl2
Passende 10 X reaksjonsbuffer sendes som oftest sammen med polymerase og med separate løsninger av 25 mM MgCl2. Mg2+-ioner danner et løselig kompleks sammen med dNTP’er som er viktig for innbygging av nucleotidene. Mg2+ ioner stimulerer i tillegg polymeraseaktivitet og øker smelte-temperaturen for DNA-dobbelheliksen og bindingen mellom primerne og DNA-matrisen. Det er nødvendig å optimalisere Mg2+ konsentrasjon. Feil konsentrasjon kan føre til tilleggsprodukter eller lavere utbytte av ønsket produkt.
DNA matrise/templat
DNA som er framstilt fra ulik type vev eller organismer kan benyttes. Man kan i tillegg kjøpe genomisk DNA fra produsent. Templatkonsentrasjonen bør ikke være for høy for å unngå at sekvenser som ikke er avgrenset av primer vokser raskere enn lineært. DNA burde i tillegg være intakt over lengden som skal amplifiseres. Dersom DNA’en er degradert kan kun kortere områder amplifiseres. Det er i tillegg viktig at inhibitorer til reaksjonen ikke er tilstede. Disse kan f.eks være detergent, EDTA eller fenol. Det er svært viktig at man har fullstendig denaturering av DNA før PCR begynner (93-95ºC i 5 min.)
Primerpar
Valg og konstruksjon av primerpar er viktig fordi primerne definerer det fragmentet man ønsker å amplifisere. For å kunne konstruere et primerpar må man kjenne DNA sekvensen av det genområdet man ønsker å klone. En rekke universiteter og forskningsinstitusjoner tilbyr gratis adgang til DNA informasjon for sekvenserte gener.
Programvaren som vi benyttet for å konstruere primere var Primer 3 som finnes på Http://www-genome.wi.mit.edu/cgi-bin/primer/primer3.
Konstruksjon av primerpar
Den optimale lengden av primerne er ca 20-30 nukleotider. I prinsippet kan man velge et hvilket som helst punkt på genet men følgende momenter bør passes på:
|
|
Alle fire basene burde være like mye representert i primerne. Dette er for å unngå at en av primerne bindes bedre enn den andre. Praktisk kontrolleres dette ved at GC % reguleres |
|
|
Primerne må ikke bestå av avsnitter som er inter- eller intramolekylær komplementær da man kan få kloningsartifakter. |
|
|
Man bør unngå DNA-sekvenser med utpreget sekundærstruktur slik at begge primerne bindes like lett. |
|
|
Ingen av primerne bør ha en 3’ T da T er den minst diskriminerende nukleotid ved 3’ posisjon og primere med en 3’ T har større sjanser for mismatch. |
|
|
Dissosiasjonstemperatur av primerparet bør være nokså likt. |
.
Primer 3
Man får, ved bruk av Primer 3 for konstruksjon av primere, mulighet til å sette en del betingelser for forskjellige egenskaper til det ferdige produktet samt begrensinger til søksområde i det aktuelle genet.
|
|
Source sequence Gensekvensen man skal bruke for å velge primere. |
|
|
Targets Mål basesekvens som man ønsker skal dekkes av minst en av primerne. Verdien har følgende notasjon: Begynnelse, lengde Eks. 1310,5 der 1310 er base i målsekvensen og 5 betyr denne basen og 4 baser rundt den. |
|
|
Excluded regions Primeroligoer kan ikke overlappe området spesifisert i målsekvensen. Man bruker samme notasjon som for "Targets". |
|
|
Produkt Size Man velger minimum, optimum og maksimum lengde i baser av PCR produkt (amplikon størrelse). |
|
|
Number to return Antall primerpar som programmet skal finne. Disse sorteres etter "kvalitet". |
|
|
Primer size Minimum, maksimum og optimum lengder i baser for en primeroligo. |
|
|
Primer Tm Minimum, maksimum og optimum smeltetemperatur i celsius for em primeroligo. Den mest brukte formelen for beregning av smelte temperatur er: Tm=(antall a+t) * 2ºC + (antall g+c) * 4ºC |
|
|
Maximum Tm Difference Maximum akseptert forskjell i smeltetemperatur mellom venstre og høyre primere. |
|
|
Product Tm Minimum maksimum og optimum smeltetemperatur for amplikonet. Primer 3 bruker følgende ligning for beregning av amplikonsmeltetemperatur: Tm=81.5 + 16.6(log10([Na+]))+.41*(%GC)-600/amplikonlengde |
Resultater av Primer konstruksjon for enzymet catalase
Primer for enzymet catalase, homo sapiens
Primer3 Output (primer3_www_results.cgi v 0.2)Primer3 Output
No mispriming library specified
Using 1-based sequence positions
LEFT PRIMER 1164 20 60.15 50.00 7.00 0.00 gcctgggacccaattatctt
RIGHT PRIMER 1366 20 59.95 55.00 3.00 2.00 gaatctccgcacttctccag
PRODUCT SIZE: 203, PAIR ANY COMPL: 4.00, PAIR 3' COMPL: 1.00
1141 ctatcctgacactcaccgccatcgcctgggacccaattatcttcatatacctgtgaactg
1201 tccctaccgtgctcgagtggccaactaccagcgtgatggcccgatgtgcatgcaggacaa
1261 tcagggtggtgctccaaattactaccccaacagctttggtgctccggaacaacagccttc
1321 tgccctggagcacagcatccaatattctggagaagtgcggagattcaacactgccaatga
Primer for enzymet catalase, oryza sativa
Primer3 Output (primer3_www_results.cgi v 0.2)Primer3 Output
No mispriming library specified
Using 1-based sequence positions
LEFT PRIMER 602 20 59.81 45.00 6.00 0.00 tgcacatatggtacccgaaa
RIGHT PRIMER 801 20 60.01 60.00 4.00 1.00 ggaactgcactagcctctgg
SEQUENCE SIZE: 4167
PRODUCT SIZE: 200, PAIR ANY COMPL: 4.00, PAIR 3' COMPL: 1.00
541 tgcaagatactttacatcagtgagtgtaaaaaatccttgccgggatcaggtgaaaaatct
601 ctgcacatatggtacccgaaaaaacaaccaatactgtctcaactggcttttgtcgtatgt
721 gtcaatgtgaacagcatctgcagcccgatcaaaaattcccattggccgtcgcgttcacat
781 tccagaggctagtgcagttccaaaaaaaaaaatcgctacgtttatctgacgagcagagag
Primer for enzymet catalase, oryza sativa, intron
Har valgt å inkludere et område kjent som intron. Det kan ofte være hensiktsmessig å velge et område med kjente introns. Man vil da være i stand til å sammenligne kjente konsentrasjoner av DNA med ukjent konsentrasjon av RNA. De amplikoner som er laget på DNA matrisen vil være større enn amplikoner av RNA.
No mispriming library specified
Using 1-based sequence positions
OLIGO start len tm gc% any primer3_www_results_help.cgi - PRIMER_THREE 3' seq
LEFT PRIMER 2908 20 59.91 55.00 2.00 2.00 ccaccacaacaaccactacg
RIGHT PRIMER 3606 20 59.63 50.00 8.00 2.00 cttgcctgcagtgcatttag
SEQUENCE SIZE: 4167
INCLUDED REGION SIZE: 4167
PRODUCT SIZE: 699, PAIR ANY COMPL: 4.00, PAIR 3' COMPL: 2.00
TARGETS (start, len)*: 3100,5
2821 gacgaggatcttctcctactccgacacgcagcgccaccgcctcggaccaaactacctgct
2881 gcttccgcccaacgcgcccaagtgcgcccaccacaacaaccactacgacggcttcatgaa
2941 cttcatgcaccgcgacgaggaggtcgactacttcccatcccgctacgatcctgccaagca
3001 cgccccccgctaccccatcccctccgccaccctcaccggccgccgcgagaaggtaattct
3061 gttgattgatttgcgtttggtttttacatttcactatattttctttcttaattactactt
3121 cccccgtttcagattataagatgttttaattttagtcaatctcaaactataagtttaact
3181 aagtttatagataaatatagtaataattacaacaccaaattagtttcattaaatcattaa
3241 atctatagttcaatatatttattatgagttaaaaatgttactattttttatataaactta
3301 gtcaaatttaaaacagtttaattttgaccaaagttaaaacgatttataatctaaaacagg
3361 agaagtagtaatagctagtcaatttattaactggttttgatcgatcgggttgcgagtgca
3421 ggtggtgattgccaaggagaacaacttcaagcagccaggggagaggtaccgttcatggga
3481 tccggcaaggtatgtacacacatgcagatgcagcaaccgaattaactgtcgagagactga
3541 actaaactgaattagtcaactagactgattgactgatgaacaaatgctaaatgcactgca
3601 ggcaagaccggttcatcaagagatggatcgacgcactctctgaccctcgcctcacccacg
tttct
målsekvensagaagt intronsekvens