ØVING 2
Bioinformatikk TES 8047
Oppgave:
Skriv litt om enzymet 3-metyladenin DNA glykosylase, finn forskjellige
organismer som har dette enzymet, utfør multippel sekvensanalyse og tegn
fylogenetisk tre.
3-Metyladenine DNA glycosylase II
Offisielt navn: DNA-3-metyladenin glykosylase II.
Alternativt navn: DNA glykosidase II.
Superfamilie: DNA glykosylase
Familie: 3-Metyladenin DNA glykosylase II (gen alkA eller aidA)
Klasse: Alpha proteiner
Folding: DNA-glykosylase multiheliks; består av to alpha-”domains”
Reaksjon som katalyseres: Hydrolyse av alkylert DNA, frigir 3-metyladenin, 3-metylguanin, 7-metylguanin og 7-metyladenin.
Det genetiske materialet i alle celler, DNA, har begrenset kjemisk stabilitet og forfaller eller skades spontant in vivo pga. hydrolyse, oksidasjon og ikke-enzymatisk metylering. For at cellene skal kunne overleve uten for høye mutasjonsfrekvenser, er de avhengige av effektiv DNA-reparasjon. Cellene har derfor utviklet spesifikke enzymatiske reparasjonssystemer som reparerer kjemiske forandringer eller skader på DNA. I baseutkuttingsreparasjonen (BER) er DNA-glykosylasene nøkkelenzymer. Disse katalyserer det første trinnet i BER. DNA-glykosylasene fjerner modifiserte eller skadde baser i DNA ved å kutte N-glykosylbindingen mellom DNA-basen og sukkeret basen er festet til (deoksyribose). Ulike DNA-glykosylaser fjerner ulike typer skader, og blant de viktigste er DNA-glykosylasene som fjerner alkylerte baser.
Alkyleringsreaksjoner fører til skader på DNA. En type alkyleringsskade er metylering av DNA-basene. Metyleringen skjer i hovedsak på N- og O-atomene til basene, og resulterer i O- og N-metyleringsprodukter som er skadelige for cellen ved at de er cytotoksiske eller gir opphav til mutasjoner. N-metyleringsproduktene er mer vanlige enn O-metyleringsproduktene, og vanlige N-metyleringsprodukter er 3-metyladenin (m3A) og 3-metylguanin (m3G) som begge er cytotoksiske. N-alkylerte baser repareres ved baseutkuttingsreparasjon, hvor basen, som tidligere nevnt, først fjernes av en DNA-glykosylase.
Bakterien Escherichia coli har to ulike DNA-glykosylaser for å fjerne N-alkylerte baser i DNA: m3A-DNA-glykosylase II og m3A-DNA-glykosylase I.
M3A-DNA-glykosylase II, også kalt AlkA, fjerner mange forskjellige alkylerte baser fra DNA, inkludert m3A og m3G. AlkA kodes av alkA-genet. Eksponering for metylerende agenter induserer AlkA-aktivitet. AlkA-proteinet består av 282 aminosyrer, har en dimensjon på 25´45´50 Å og en molekylvekt på 31400. Proteinet er bygget opp av tre "domains". "Domain 1": en blandet a-b-struktur.
"Domain 2": en bunt av syv a-helikser.
"Domain 3": fire a-helikser.
Det aktive setet er lokalisert i kløften mellom "domain 2" og "domain 3". Kløften er rik på elektron-donerende aromatiske sidekjeder som er godt egnet til å danne p-donor/akseptor interaksjoner med elektron-fattige metylerte baser. AlkA kan dermed kutte glykosylbindingen til de modifiserte nukleosidene lokalisert i denne kløften når de samtidig har C(1')-atomet nær den katalytisk viktige Asp-238 i bunnen av kløften.
M3A-DNA-glykosylase I, også kalt Tag, fjerner spesifikt m3A. Molekylvekten er 21100. Tag uttrykkes kontinuerlig.
AlkA og Tag har ingen signifikant sekvenshomologi, til tross for funksjonell likhet (begge fjerner m3A).
Det er funnet DNA-glykosylaser hos en mengde forskjellige organismer fra bakterier til mennesker, og det viser seg at disse alkyleringsreparasjonsenzymene har en ganske heterogen struktur. Den mammalske alkylbase-DNA-glykosylasen har ikke signifikant sekvenshomologi med hverken Tag eller AlkA, selv om den, som AlkA, fjerner en rekke forskjellige alkyleringsprodukt. Saccharomyces cerevisiae sin m3A-DNA-glykosylase (kodet av MAG-genet) har derimot homologi med AlkA. Mens mellom plante-enzymer og AlkA er det ingen sekvenshomologi; Arabidopsis thaliana N-glykosylase Amag ligner mer på det mammalske enzymet.
Organismene jeg valgte:
Homo
sapiens
Mus
musculus
Escherichia
coli
Archaeoglobus
fulgidus
Rattus
norvegicus
Chlamydophila
pneumoniae
Bacillus
subtilis
Pyrococcus
abyssi
Arabidopsis
thaliana
Saccharomyces
cerevisiae
Multippel sekvensanalyse
For å sammenligne 3-metyladenin DNA glykosylaser fra forskjellige organismer
utførte jeg en multippel sekvensanalyse ved hjelp av ClustalX.
Hvis du vil vite mer om multippel sekvensanalyse, kan du klikke her.
Resultatet av sekvensanalysene:
ClustalX
Multippel sekvensanalyse
ClustalX Multippel
sekvensanalyse der sure og basiske aminosyrer er fargelagt
Multippel sekvensanalyse der
sure og basiske aminosyrer er fargelagt (html-dokument)
Multippel
sekvensanalyse der hydrofobe og hydrofile aminosyrer er fargelagt
(html-dokument)
Fylogenetisk tre
Resultatene fra sekvensanalysene ble brukt til å konstruere fylogenetisk
diagram/tre ved hjelp av Drawgram/Drawtree:
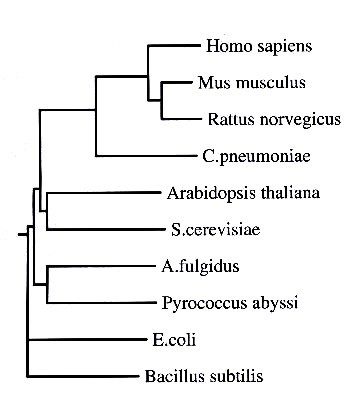
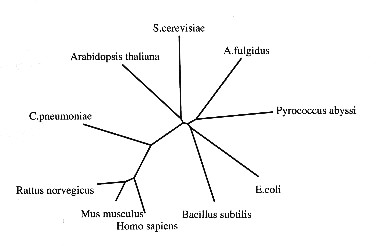
Fylogenetisk diagram for 3-metyladenin DNA glykosylase Fylogenetisk tre for 3-metyladenin DNA glykosylase
Hvis du vil lese om utførelsen av øving 2, kan du klikke her